El proyecto del genoma humano en la balanza
Miguel Ángel Cevallos
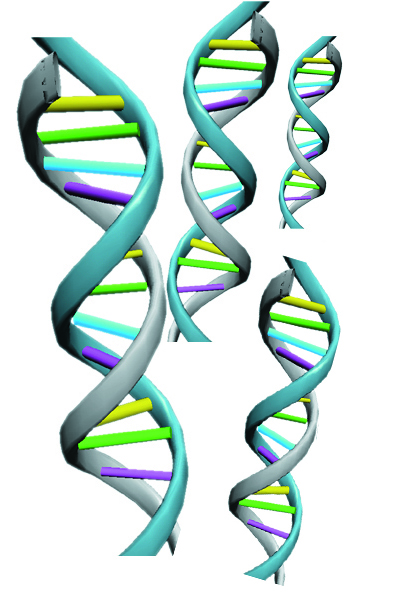
Ilustración: Carlos Durand
Cuando se concluya la secuencia del genoma humano, nuestras vidas cambiarán de un modo tan profundo como con el descubrimiento de la energía atómica y el uso masivo de computadoras. Es evidente que no todos los cambios serán para bien, pero, desde luego, habrá enormes beneficios.
No creo que alguien pueda imaginarse lo que significa pasar toda la vida dentro de una burbuja. Sin embargo, para los niños con inmunodeficiencia combinada severa esa condición es una realidad. Estos niños sufren de una enfermedad genética que impide que su sistema inmunológico se desarrolle; es decir, sus cuerpos carecen de defensas contra los organismos patógenos por lo que se les debe proporcionar un ambiente totalmente estéril en el cual se encuentran aislados, no sólo de los organismos patógenos sino de cualquier contacto humano. Aún así, viven poco tiempo.
Existen varios tipos de inmunodeficiencia combinada severa, todos de carácter hereditario. Los individuos afectados portan algún gene defectuoso que impide que su sistema inmunológico se desarrolle en forma adecuada; por ejemplo, en la inmunodeficiencia combinada severa -X1 el gene defectuoso (gamma-c) se localiza en el cromosoma X, y evita que se formen los linfocitos, células centrales del sistema inmune.
abril surgió una esperanza para estos niños. Los investigadores de un equipo francés lograron corregir el defecto: tomaron células "madre" de la medula espinal, que dan origen a los linfocitos, de un grupo de niños enfermos. Con técnicas de ingeniería genética introdujeron a estas células un gene normal gamma-c, para sustituir las funciones del gene defectuoso y luego se las regresaron a los niños enfermos. Transcurridos diez meses, éstos tenían ya niveles normales de linfocitos, pudieron abandonar sus "burbujas" y llevar una vida normal. Tales "milagros" de la tecnología médica de hoy son un ejemplo de lo que será la medicina en un futuro cercano gracias al proyecto del genoma humano.
Las metas
Por muchas hojas que escriba nunca serán las suficientes para ponderar, de manera adecuada, las implicaciones de conocer en detalle el genoma humano. Sin embargo, antes que nada debe quedar claro cuál es la meta del proyecto: lo que pretende es determinar el orden secuencial de los aproximadamente tres mil millones de nucleótidos que conforman el adn de nuestros 23 pares de cromosomas. En estos tres mil millones de nucleótidos se espera identificar algo así como 100 000 genes que, a final de cuentas, son los fragmentos de adn que portan las instrucciones para todas y cada una de nuestras funciones biológicas. Por extraño que parezca, esos 100 000 genes sólo representan alrededor de un 5% del adn; hasta donde sabemos, el 95% restante no tiene ninguna función.
Obviamente una parte integral del proyecto del genoma humano consiste en desarrollar las herramientas de cómputo que se requieren para analizar y organizar la información que se obtenga de la secuencia del adn y, desde luego, estudiar las implicaciones éticas, legales y sociales que surjan de tener en nuestras manos esa información.
 Los seres humanos contamos con 23 pares de cromosomas en cada una de nuestras células. Veintidós pares de éstos son iguales entre hombres y mujeres, sin embargo, los hombres cuentan con un cromosoma X y otro Y; las mujeres cuentan con dos del tipo X.
Los seres humanos contamos con 23 pares de cromosomas en cada una de nuestras células. Veintidós pares de éstos son iguales entre hombres y mujeres, sin embargo, los hombres cuentan con un cromosoma X y otro Y; las mujeres cuentan con dos del tipo X.
Avances
En 1986 el doctor David Smith, director del Programa del Genoma Humano del Departamento de Energía de los Estados Unidos, recibió muchas críticas cuando anunció que se estaba iniciando un proyecto de tal calibre. Para muchos, secuenciar todo el adn humano era extremadamente descabellado dada su complejidad técnica y la falta de herramientas para analizar los resultados. Sin embargo, el Instituto Nacional de Salud de ese país pronto se interesó en el proyecto, formó su propio programa y unió fuerzas con el Departamento de Energía. Hoy en día, el proyecto del genoma humano abarca universidades e instituciones de investigación de muchas partes del mundo. Entre las más importantes fuera de los Estados Unidos se encuentra el Instituto Sanger del Reino Unido. Catorce años después, los logros tanto técnicos como científicos son sorprendentes: en 1986, cuando un investigador quería determinar una secuencia de adn lo tenía que hacer con un método largo y tedioso, para obtener en un par de días, el orden de algo así como 500 nucleótidos. En la actualidad, existen máquinas automatizadas que pueden determinar 850 nucleótidos de la secuencia de 96 muestras diferentes, al mismo tiempo, en dos horas, y pueden trabajar sin interrupción a este ritmo las 24 horas del día. También se han desarrollado bancos de datos de secuencias de adn que pueden consultarse desde cualquier parte del mundo a través de Internet. Estos bancos de datos se actualizan día a día y ofrecen herramientas de cómputo a los investigadores para analizar las secuencias de adn de su interés, de modo totalmente gratuito. El proyecto del genoma humano ha avanzado a tal velocidad que se espera esté terminado para el 2003, dos años antes de lo planeado. De hecho, la secuencia del cromosoma 22 se publicó en la revista Nature en diciembre de 1999, y la del cromosoma 21 en mayo de este mismo año.
 Representaciones de la estructura molecular del ADN
Representaciones de la estructura molecular del ADN
Genes, empresas y patentes
No todo es miel sobre hojuelas para los investigadores que participan en el consorcio multinacional del proyecto del genoma humano. En mayo de 1998, J. Craig Venter, un científico audaz y visionario, anunció que su compañía, Celera Genomics, armada con cerca de 300 máquinas automatizadas de secuencia y con equipo de cómputo de última generación, determinaría la secuencia del genoma humano en tres años y a menor costo: no sólo cumplió sus promesas, sino que en el congreso "Genomes 2000" realizado en París, el pasado abril, anunció que la tarea estaba concluida, aún cuando no mostró sus datos. Sin embargo, Celera promete compartir la información con cualquier científico, a condición de que no se la dé a otros, y que si hay un descubrimiento por el que se pueda obtener una patente, su compañía debe gozar de una participación en la misma. Esto marca una diferencia irresoluble con los científicos del proyecto del genoma humano, cuyos resultados se hacen públicos casi tan rápidamente como se obtienen, impidiendo así que sean sujetos de patente.
Pero detengámonos un momento, ¿los genes se pueden patentar del mismo modo que si se tratara de un nuevo chip de computadora? Si es así, ¿por qué no pueden patentarse las vacas?
La respuesta no es trivial. Cualquier cosa patentable debe tener algunas propiedades básicas: el invento debe ser importante, novedoso y útil. La vaca no cumple con algunos de estos requisitos: obviamente es útil, pero ya sabemos para qué sirve y cómo se usa. En un primer examen, diríamos que un gene del cual no sepamos para qué sirve y cómo se podría usar no es sujeto de patente. Sin embargo, hay otros genes sobre los cuales puede indicarse claramente para qué sirven y cómo podría aprovecharse este conocimiento. Pongamos por caso el gene para la fibrosis quística. Se ha descubierto que este gene tiene las instrucciones para producir una proteína defectuosa, involucrada en el transporte del ion cloro en nuestras células; el hallazgo permitió diseñar un método de diagnóstico que distingue el gene defectuoso de su contraparte normal y se utiliza para identificar a los individuos enfermos y a los portadores. Este gene también podría usarse para desarrollar una terapia genética eficaz. Ahora bien, el trabajo de investigación para identificarlo fue complejo y muy costoso. El poder patentar este descubrimiento permite recuperar el dinero invertido y, desde luego, esperar ganancias: si no existiera el aliciente económico, las compañías farmacéuticas no estarían interesadas en realizar este tipo de investigaciones. Además, hay que tener en cuenta que son muchos los proyectos en los que se invierte, y muy pocos los que llegan a la fase de mercado. Las industrias privadas son las responsables de muchos de los desarrollos biotecnológicos más importantes de los últimos años; la participación en ellos de las universidades y los institutos de investigación es cada vez menor, debido a su alto costo.
La oficina de patentes de los Estados Unidos todavía no tiene una posición muy clara sobre cuáles genes son patentables y cuáles no. Mientras tanto, las compañías biotecnológicas, como Celera Genomics o como Incyte Pharmaceuticals, han inundado la oficina de patentes con solicitudes para la secuencia de miles de segmentos de adn, utilizando el argumento de que algún día podrían utilizarse para localizar o identificar algún gene responsable de un mal hereditario, pero sin especificar de cuál enfermedad se trata ni cómo identificarla. Cuando se les pide que expliquen cuál es la función del gene, lo que suelen presentar es un análisis en computadora de su posible función basada en su similitud con otros genes descritos, sin mostrar nunca una prueba experimental. Si se permite patentar cualquier segmento de adn, se puede desalentar a otros investigadores a que se interesen en ese gene, puesto que si se hace algún descubrimiento importante, parte de los derechos de la posible patente pertenecerán a la compañía que patentó la secuencia correspondiente.
La posición del consorcio del proyecto del genoma humano es que sólo deben patentarse aquellos genes de los cuales se tenga una descripción y un posible uso claro y preciso. Los integrantes de ese proyecto proponen que la información cruda de la secuencia debe depositarse inmediatamente en bases de información de dominio público, con el fin de que pueda ser utilizada por cualquiera (Acuerdo de Bermudas). Esta posición la defienden el presidente Clinton de los Estados Unidos y el primer ministro Blair del Reino Unido. Cualquier decisión que se tome en este sentido definirá, en gran medida, el futuro de la biotecnología.
 Cada color corresponde a un nucleótido diferente. La adenina siempre se une a la timina y la citosina a la guanina.
Cada color corresponde a un nucleótido diferente. La adenina siempre se une a la timina y la citosina a la guanina.
Usos y abusos de la información Genética
Cuando se concluya la secuencia del genoma humano, nuestras vidas cambiarán de un modo tan profundo como con el descubrimiento de la energía atómica y el uso masivo de computadoras. Es evidente que no todos los cambios serán para bien, pero, desde luego, habrá enormes beneficios que no siempre son fáciles de imaginar. Uno de los cambios que constataremos más pronto es que podremos diagnosticar de un modo certero muchas de las aproximadamente 3 000 enfermedades de origen hereditario que nos aquejan. También tendremos la capacidad de evaluar la predisposición a adquirir males que tengan algún componente genético, por ejemplo la predisposición a acumular colesterol. En un futuro cercano habrá terapias genéticas que podrán corregir el problema de ciertas enfermedades hereditarias que son resultado de la presencia de un solo gene defectuoso: hago esta aclaración porque hay muchas enfermedades hereditarias que sólo se manifiestan cuando heredamos no sólo uno sino varios genes defectuosos, y las estrategias de terapia genética que se están desarrollando todavía son incipientes para contender con más de un gene al mismo tiempo
Una vez secuenciados los 100 000 genes que se calcula que tiene el ser humano, se podrán diseñar mejores fármacos. Tomemos, por ejemplo, el caso de algunos de los nuevos analgésicos. Se sabía que la aspirina (ácido acetil-salicílico) elimina el dolor porque se pega a una proteína llamada ciclo-oxigenasa 2 (COX2) evitando que funcione. Esta proteína es parte esencial de la cadena de eventos que nos permite sentir dolor. Pero también se sabe que la aspirina puede causar úlceras y gastritis lo cual, por cierto, nada tiene que ver con que la aspirina sea ácida, sino más bien con que inhibe la producción del moco que recubre el estómago. Este desagradable efecto se debe a que la aspirina se pega a otra ciclo-oxigenasa (COX1) muy parecida a la COX2 pero que participa en la cadena de eventos que nos conduce a producir el moco que recubre la pared del estómago. Una vez que se pudo determinar la secuencia de los genes de las proteínas COX1 y COX2, también fue posible producir las proteínas en gran escala y se definió su estructura en finísimo detalle. Con estos datos fue factible diseñar un fármaco que sólo se pega a COX2 y no reconoce a COX1. De este modo tenemos analgésicos que quitan el dolor y no tienen los efectos colaterales indeseados. Estos nuevos analgésicos están a la venta, incluso en el mercado mexicano.
La información genética que ya tenemos en nuestras manos se está usando de muy diversos modos. Un ejemplo es el de la comunidad judía ortodoxa ashkenazi de Nueva York. En esta comunidad aparece con mucha frecuencia el llamado Síndrome de Tay-Sachs, una enfermedad que se produce porque los afectados no pueden degradar unas sustancias esenciales del sistema nervioso llamadas gangliósidos. Los primeros síntomas aparecen antes de cumplir un año de edad: los enfermos presentan un grave retraso en su desarrollo y tienen dificultad para alimentarse. Poco tiempo después quedan ciegos y suelen morir antes de los tres años de edad. Esta enfermedad aparece en uno de cada cuatro embarazos en que ambos padres son portadores del gene para el Tay-Sachs. Para evitar que el terrible mal siguiera afectando a su comunidad, los ashkenazi neoyorquinos instrumentaron un ingenioso programa, en el cual cada año se visita a las escuelas preparatorias de la comunidad judía y se ofrece detectar a aquellas personas que son portadoras del gene de Tay-Sachs. A los inscritos en el programa se les asigna un número confidencial que los identifica, y se les hace el diagnóstico. En última instancia, la única información que tiene el comité es un número y un diagnóstico del gene de Tay-Sachs asociado a ese número. Cuando una pareja de la comunidad quiere casarse entrega al comité su número confidencial y éste les informa si constituyen una pareja con riesgo o no. Con estas medidas han logrado disminuir casi por completo la incidencia de esta enfermedad. El programa ha tenido tanto éxito que ya se piensa en aplicarlo a otras enfermedades que aquejan a la comunidad ashkenazi, como es el caso de la fibrosis quística.
 El proceso de secuenciación implica fragmentar el ADN; la máquina secuenciadora
lee cada segmento y saca el orden de los nucleótidos.
El proceso de secuenciación implica fragmentar el ADN; la máquina secuenciadora
lee cada segmento y saca el orden de los nucleótidos.
¿De quién y de dónde?
Muchísimas personas tienen la inquietud de saber de quién es el genoma que se secuenció: ¿de Tony Blair?, ¿de William Clinton?, ¿o acaso el de Tom Cruise? No hay respuesta o, más bien, se siguió un mecanismo para que ni los mismos científicos pudieran conocerla. Se pidió a un grupo grande de donadores anónimos, de varias razas y de ambos sexos, que cedieran unos cuantos mililitros de sangre; a los hombres, se les solicitó un poco de semen. Posteriormente, se aisló adn de 20 muestras elegidas al azar, se mezclaron y precisamente esta mezcla fue la que secuenció el consorcio multinacional del proyecto del genoma humano. En la compañía Celera Genomics usaron otra estrategia: determinaron la secuencia del adn de un solo individuo, cuya identidad permanecerá en el anonimato (aunque las malas lenguas dicen que se trata del mismísimo doctor Venter).
La muestra para aislar el adn pudo haberse tomado de cualquier tejido ya que, con poquísimas excepciones, todas las células de nuestro cuerpo tienen la misma información genética. Se eligió semen por dos razones: primero, definitivamente la toma de la muestra está lejos de ser dolorosa y segundo, los espermatozoides contenidos en el semen contienen sólo 23 cromosomas, en comparación con los demás tipos de células que tienen por duplicado cada uno de los 23 cromosomas. Las mujeres donaron sangre porque de este tejido es muy fácil aislar adn. Los óvulos contraparte de los espermatozoides, son células que sólo tienen 23 cromosomas, pero su uso se descartó por la dificultad y peligro que encierra tomar las muestras.
Cada especie animal o vegetal tiene un número de cromosomas que lo caracteriza, por ejemplo, el perro tiene 40 cromosomas, el chimpancé 44 y la mosca de la fruta 4. Los seres humanos contamos con 23 pares de cromosomas en cada una de nuestras células. Veintidós pares de estos cromosomas son iguales entre hombres y mujeres, sin embargo, los hombres cuentan, además, con un cromosoma X y otro Y. Las mujeres, en contraste, cuentan con dos del tipo X.
Los 46 cromosomas de cada una de nuestras células se encuentran cuidadosamente empacados en un minúsculo organelo celular que se llama núcleo. Cada uno de nuestros cromosomas se compone químicamente de una larguísima molécula de adn y de proteínas que se asocian de una manera muy estrecha. La molécula de adn es la que contiene la información genética codificada. Las proteínas protegen al adn y ayudan a que se interprete en los tiempos y los modos adecuados.
En general, el manejo del diagnóstico genético es un asunto delicado pero en ocasiones es simple, claro y beneficioso. Ése es el caso de la fenilcetonuria o la hematocromatosis, enfermedades hereditarias que causan trastornos en el metabolismo y para las cuales existen terapias simples y efectivas; la única condición para que funcionen es que se inicien desde el nacimiento. Por eso, un diagnóstico genético temprano y certero permitirá que los afectados tengan una vida normal.
En enfermedades mortales de desarrollo más o menos rápido, como la enfermedad de Tay-Sachs o la fibrosis quística, el diagnóstico genético no beneficia al enfermo, y en realidad sólo se utiliza para confirmar el diagnostico médico hecho por otros medios. Quizá el beneficio más claro sea que las familias de individuos afectados podrán someterse a un diagnóstico genético para reconocer cuáles de sus miembros son portadores.
El manejo del diagnóstico genético puede ser mucho más complicado cuando se trata de enfermedades extremadamente discapacitantes, como la Corea de Huntington, la ataxia espinocerebral o la enfermedad de Lou Gehrig, cuyos primeros síntomas aparecen cuando el individuo afectado ya es un adulto. En estos casos, un diagnóstico genético previo a la aparición de los primeros síntomas, ayuda poco a los enfermos, puesto que no existen terapias efectivas que retrasen o contrarresten a la enfermedad. Más aún, dado el diagnóstico, es muy probable que las compañías de seguros médicos nieguen al afectado cobertura médica, puesto que no amparan tratamientos para "condiciones" o "males" preexistentes. En estos casos, el acceso al diagnóstico genético debe evaluarse cuidadosamente, dadas las repercusiones que pudiera tener en la calidad de vida de un individuo antes de que aparezcan los primeros síntomas de la enfermedad.
Existen otros casos que vale la pena mencionar: hay defectos genéticos que lo que hacen es predisponer, más no condenar, a un individuo a sufrir una enfermedad.
Pongamos por caso el cáncer de ovario y de seno: en la población general, alrededor del 16% de las mujeres adquirirá en algún momento de su vida alguno de estos cánceres. Sin embargo, en algunas familias existen múltiples casos de mujeres afectadas por esta enfermedad. En ellas se ha descubierto la presencia de los genes BRCA1 o BRCA2, condición que aumenta hasta en 85% sus posibilidades de adquirir alguno de estos cánceres a lo largo de sus vidas. La mujer a la cual se le detecta la presencia de los genes BRCA1 o BRCA2 no está condenada a morir de cáncer, incluso puede que nunca llegue a adquirir esta enfermedad; sin embargo, una mujer que se sepa portadora de alguno de estos genes está en condiciones de decidir si se hace exámenes médicos más a menudo o ser mucho más radical, y hacer que le eliminen quirúrgicamente los ovarios y los senos. Ahora bien, la información de carácter genético, aunque sea confidencial, nos lleva otra vez a los problemas de los seguros médicos: para solicitar a una compañía aseguradora que pague una operación preventiva de este tipo, la afectada debe indicar su predisposición genética, en cuyo caso, por tratarse de una condición preexistente, la compañía le negará los recursos económicos. En países donde el sistema médico se maneja, en gran medida, con base en seguros médicos, la confidencialidad de la información genética es esencial para poder tener acceso a un buen servicio médico. Ahora, como bien dice mi amigo Mario Zurita, quizá a la larga, la información genética será tan cotidiana, que nos daremos cuenta que todos tenemos genes defectuosos y propensiones genéticas a diversas enfermedades. ¡Ni Cindy Crawford tendrá un genoma perfecto!
Aún así, tenemos que reinventar una nueva manera de concebir los seguros médicos para que no queden desprotegidas las personas con este tipo de problemas. En México el asunto aún no es tan importante, puesto que la mayor parte de los servicios médicos dependen del sistema de gobierno y no de los seguros. Sin embargo, éstos no son los únicos aspectos que hay que cuidar, es necesario legislar sobre el uso de la información genética, ya que dicha información no sólo podría utilizarse para negar coberturas de seguros médicos sino, con una visión racista, también para negar trabajo o visas.

Mensajes moleculares
Los primeros en entender cómo es la estructura fina de una molécula de adn fueron James Watson y Francis Crick y por ese trabajo se les concedió el Premio Nobel de Medicina y Fisiología en 1962. Una molécula de adn la podemos representar como dos cadenas que se entrelazan, una en la otra, para formar una doble hélice. Los eslabones de cada cadena son lo que llamamos nucleótidos y los hay de cuatro tipos: la adenina (A), la citosina (C), la guanina (G) y la timina (T). Las dos cadenas de nucleótidos se unen entre sí a través de enlaces químicos que se establecen entre los nucleótidos de una cadena con los nucleótidos de la otra, pero siguiendo una regla: sólo pueden establecerse enlaces químicos correctos cuando una adenina se encuentra con una timina de la otra cadena de nucleótidos, y cuando una guanina se encuentra con una citosina. Tan rigurosa es esta regla que si conocemos la secuencia de nucleótidos de una de las cadenas, podemos deducir, sin ambigüedad, la secuencia de la otra cadena. La secuencia de nucleótidos es un mensaje molecular que utiliza la célula para realizar una función en un tiempo y situación determinados. Cada mensaje "codificado" en el adn se conoce con el nombre de gene. Y lo más interesante de todo es que el código que utiliza una bacteria para guardar sus mensajes en el adn, es el mismo código genético que utilizan el caballo, el helecho o la hormiga. Lo que cambia son los mensajes, pero el código es el mismo. Afortunadamente, hace más de 30 años que desciframos el código genético, de tal suerte, que si conocemos la secuencia de nucleótidos de una molécula de adn de cualquier organismo podemos conocer el mensaje que está inscrito en ella.
De forma breve, para secuenciar el genoma de un organismo, primero se purifica el adn y se rompe en pedacitos. Cada pedazo de adn se somete a una reacción bioquímica que utiliza una enzima llamada adn polimerasa. Esta enzima lo que hace es una copia complementaria de una de las cadenas del pedazo de adn elegido, pero en lugar de utilizar sólo nucleótidos normales (los eslabones de la cadena) también utiliza cuatro nucleótidos modificados. La modificación consiste en que cada nucleótido modificado tiene un "colorante" pegado que se puede "leer" con una cámara digital acoplada a un rayo láser. Digamos, rosa para las T, amarillo para las G, azul para las C y verde para las A. Después de la reacción, tenemos en conjunto copias de la cadena de adn que se quiere secuenciar, pero con un nucleótido "coloreado" en cada posición del adn. Lo que a final de cuentas hace el lector es detectar qué color hay en cada posición y así determinar la secuencia del adn deseado.
La tentación de la eugenesia
"Eugenesia" puede parecer una palabra rara, pero tiene más de 100 años en uso; su significado viene de las raíces griegas que quieren decir "bien nacido". El término fue acuñado por Francis Galton, un sobrino de Darwin, quien propuso que se deberían poner en práctica nuestros conocimientos de cómo opera la evolución y generar programas gubernamentales para crear una "mejor" raza humana. Este tipo de ideas tuvieron muchos adeptos; en países como la Alemania nazi o en los Estados Unidos se crearon institutos de investigación y programas gubernamentales con ese propósito. Millones de personas tuvieron que pagar las consecuencias: se esterilizó a homosexuales, a criminales y a retrasados mentales. Se prohibió la inmigración por miedo a "contaminar" el patrimonio genético de un país e incluso se prohibieron los matrimonios interraciales. Qué decir del genocidio nazi cometido contra la comunidad judía.
Seguramente hay muchas personas que piensan que la información que se obtenga del proyecto del genoma humano, junto con los conocimientos que tenemos hoy de genética y de biología molecular, será utilizada en programas de "mejoramiento genético" o en la planeación de la sociedad con base en el patrimonio genético de las personas. Dejando a un lado todos los aspectos éticos que están íntimamente ligados al uso de la información genética, científicamente no se puede planear a largo plazo una "mejor raza humana", por mucho que así lo deseen los grupos supremacistas de cualquier país. Ésta es una lección que nos ha costado mucho aprender y hay numerosos ejemplos para mostrarlo: en todas aquellas comunidades humanas que favorecen los matrimonios en su interior y desfavorecen los matrimonios con individuos fuera de ésta, la incidencia de enfermedades de carácter hereditario es notablemente mayor que en el resto de la población. Ya hablamos de la comunidad ashkenazi y de sus problemas de Tay-Sachs y fibrosis quística (y no son los únicos problemas de carácter genético a los que se enfrentan). Otra más es la comunidad "Nueva Germania" en Paraguay, fundada con "germanos de raza pura" por Elizabeth Nietzsche (hermana del célebre filósofo) con la finalidad de generar una superraza: hoy en día sus habitantes sufren de muchas enfermedades de carácter hereditario. No hay que extrañarse, esto no sólo sucede en los grupos humanos, también en los animales y las plantas domésticas. Pensemos en nuestras razas de perros: hermosos, rápidos y quizá con olfato magnífico, pero con enorme propensión a diversos tipos de cánceres, malformaciones, ceguera y otras enfermedades de carácter hereditario. Lo mismo ocurre con las plantas domesticadas: las variedades de maíz, trigo o arroz, producto de la llamada revolución verde, producen muchísimos más granos que las variedades silvestres, pero son extremadamente más sensibles a las enfermedades, a tal grado que tienen poquísimas probabilidades de sobrevivir si no interviene la mano del hombre.
Aunque pudiéramos construir individuos sin "genes defectuosos", a la larga sería imposible determinar qué conjunto de genes serían evolutivamente más exitosos, puesto que el propósito último en la evolución es permitir que los individuos tengan descendencia que pueda sobrevivir y reproducirse, en unas condiciones dadas del medio ambiente. Como no puede predecirse cómo será el medio ambiente en 100 000 años (bueno, ni siquiera en un año), tampoco podemos predecir cuáles genes o características serán más útiles para sobrevivir y cuáles no lo serán. Lo que es claro es que cuanto mayor sea la riqueza en diversidad genética de una población, muchas más serán sus probabilidades de sobrevivir.
 Cromosomas.
Cromosomas.
Algunas repercusiones
Los avances tanto metodológicos como conceptuales logrados durante el desarrollo del proyecto del genoma humano están cambiando nuestra manera de concebir los fenómenos biológicos. En 1996 se reportó por primera vez la secuencia completa de adn de un organismo: la bacteria Haemophilus influenzae. El trabajó no sólo sorprendió por su magnitud y calidad, sino también por el hecho de que fue una compañía biotecnológica quien lo realizó, y a una velocidad nunca antes vista. Cuatro años después, se conoce la secuencia de los genomas de cerca de 30 bacterias y se está determinando la secuencia de otras tantas más. También conocemos la secuencia completa de los genomas de organismos más complejos, como son el de la levadura, el de un gusano (Caenorhabditis elegans) y el de la mosca de la fruta, y en este mismo año tendremos la de Arabidopsis thaliana, la planta favorita de los biólogos moleculares. Además, se están determinando las secuencias de los genomas del ratón y del perro, las cuales tienen grandes similitudes con las del humano. De la comparación de estos genomas se podrán resolver cuestiones como: ¿cuáles son los genes que tenemos en común todos los organismos vivos?, ¿qué conjunto de genes hacen que una mosca sea una mosca y una planta una planta?, ¿cuál es el mínimo grupo de genes que se requiere para la vida?, ¿cuáles son nuestros parentescos evolutivos? y muchas más que sólo ahora hemos podido plantearnos.
El conocimiento de todos los genes de un organismo ha permitido que los científicos estén desarrollando métodos para determinar cuáles genes están funcionando en un órgano y cuáles no. Con estas mismas técnicas se están comparando tumores con sus contrapartes de tejido normal. Gracias a este tipo de información, y por primera vez, estamos obteniendo una visión global de las funciones celulares en condiciones normales y patológicas. Esta visión será un elemento fundamental de la mejor comprensión no sólo de la fisiología normal de las células, sino también de cómo y por qué se produce un tumor y, desde luego, proponer terapias más efectivas. La única manera de sopesar correctamente las nuevas tecnologías es estar bien informado.
Miguel Ángel Cevallos
es doctor en investigación biomédica básica. Actualmente trabaja en el Centro de Investigación sobre Fijación del Nitrógeno de la UNAM.