Avatares químicos. En busca de nuevos fármacos
Cynthia Fernández, Humberto Mendoza y José Correa
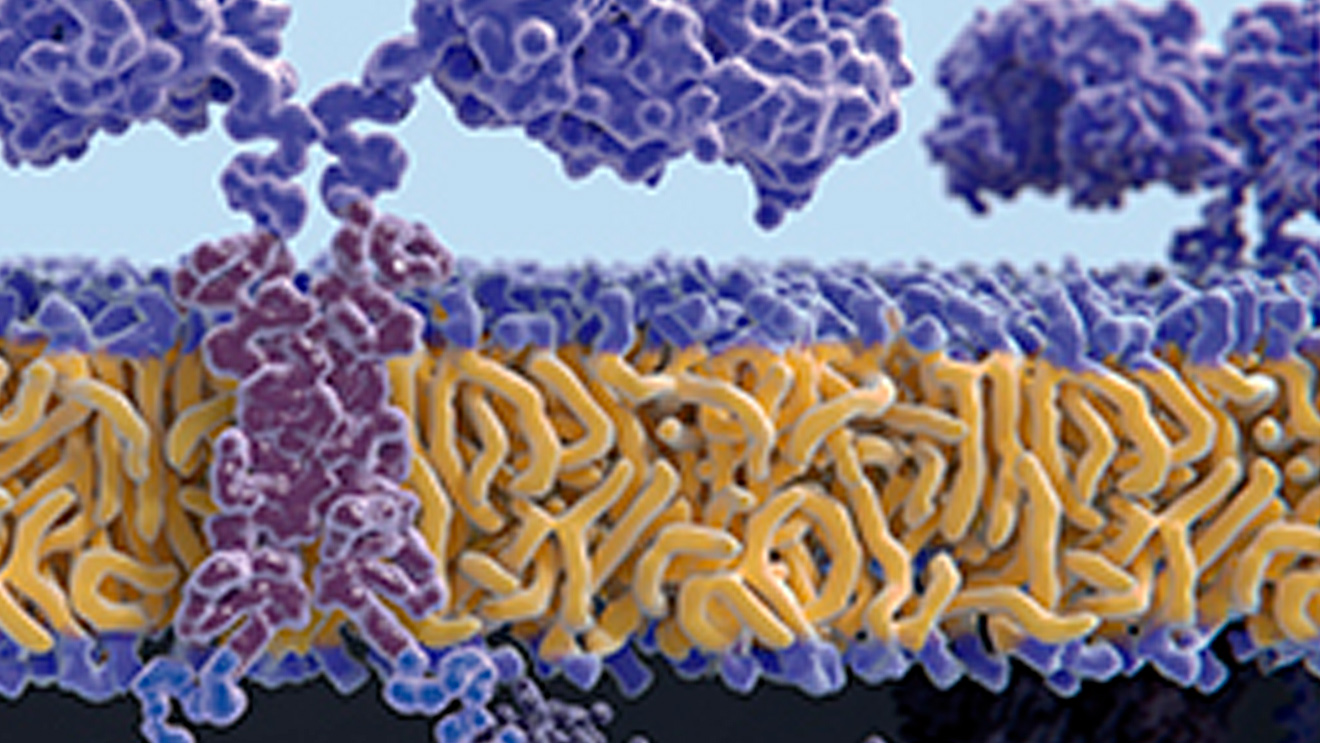
Imagen: ¿Cómo ves?
La rápida evolución de las enfermedades nos obliga a desarrollar tratamientos más eficaces en tiempo récord. Hoy, con herramientas computacionales, no solo podemos encontrar nuevas moléculas para producir medicamentos, también podemos seleccionar las más potentes y específicas y las menos tóxicas.
En el ámbito de la informática un avatar es la representación gráfica de una persona en un en un entorno virtual. Nuestro avatar puede interactuar y hacer cosas que no haríamos en la realidad. Hay avatares hasta para las mascotas. Pues bien, hoy, con las herramientas computacionales que nos permiten simular fenómenos físicos y químicos, también podemos construir representaciones tridimensionales de las moléculas de las sustancias medicinales: avatares químicos que nos ayudan a encontrar nuevos fármacos.
Mecanismo de acción
Los fármacos son compuestos químicos usados para diagnosticar, prevenir o tratar enfermedades. En clase de química se dibujan como figuras geométricas y líneas interconectadas con letras que simbolizan los elementos químicos. Pero más allá del lápiz y el papel, el avatar de estas figuras, su representación en tercera dimensión, nos permite ver el orden en el que los átomos se unen, su distribución en el espacio y cómo interactúa la sustancia con otras moléculas; todo ello se simula utilizando cálculos matemáticos.
De la distribución tridimensional de los átomos depende el efecto biológico de la molécula; es decir, su capacidad para modificar las funciones de las células del organismo. En este contexto a la molécula se le denomina ligando.
Para modificar esas funciones, el ligando tiene que adherirse primero a un elemento de la superficie de la célula: una proteína que llamamos “receptor” y también “objetivo farmacológico”. Como resultado de esa unión, se activan o bloquean funciones específicas de la célula. Esto se conoce como mecanismo de acción farmacológica.
Figuras en la arena
De los granos de arena surgió buena parte de la tecnología informática. El silicio, principal componente de la arena, es el segundo elemento más abundante en la Tierra y el primero en nuestras computadoras. Por eso a las investigaciones químicas y biológicas realizadas por medio de simulaciones por computadora se les llama estudios in silico, o en silicio, por analogía con los estudios in vivo (con organismos vivos) e in vitro (con células en tubos de ensayo y cajas de Petri).
En la farmacología clásica, las sustancias potencialmente medicinales se extraían, por ejemplo, de alguna planta, y sus efectos se probaban en tejidos o en animales vivos. Luego se buscaba el componente responsable de la actividad biológica: el principio activo. Después esta molécula se aislaba para estudiar su mecanismo de acción. Esta estrategia experimental es altamente costosa y tardada.
Los métodos de diseño de fármacos in silico van al revés de la farmacología clásica. En vez de empezar con una sustancia cuyos efectos se quieren probar, primero se selecciona una proteína receptora en la célula (un objetivo farmacológico) y luego se buscan moléculas capaces de unirse a ella. También podemos partir de una molécula específica y buscar la proteína receptora de la célula con la que pueda interactuar.
Ante todo, estructura
Todas las estrategias del diseño in silico exigen que conozcamos la estructura tanto de las moléculas de los fármacos potenciales como de las proteínas de la superficie de la célula. Estas estructuras se determinan por medio de técnicas experimentales como la cristalografía de rayos X —que se usó para revelar la estructura del ADN (veáse ¿Cómo ves?, núm. 168)— y la resonancia magnética nuclear, que toman una especie de fotografía bidimensional o tridimensional de las estructuras.
Las imágenes estructurales se convierten en números, que es como si tomáramos las medidas de una persona y las guardáramos en un archivo para construir su avatar digital. La información se almacena en bases de datos como DrugBank, Protein Data Bank, PubChem y ChEMBL. Los programas bioinformáticos utilizan estas bibliotecas virtuales para construir modelos matemáticos que calculan la forma en que pueden unirse el ligando y el receptor.
Ensayo y error
El imatinib es un medicamento que ha revolucionado la terapia contra uno de los tipos de leucemia (cáncer de células sanguíneas), la leucemia mieloide crónica. Su objetivo farmacológico es la proteína BCR-ABL, una especie de Frankenstein molecular producto de la fusión de los cromosomas 9 y 22. Esta proteína, también conocida como cromosoma Filadelfia, es una clase de cinasa, es decir una enzima que enciende y apaga procesos celulares; el problema es que dispara la proliferación desbocada de las células mieloides.
Lo que hace el imatinib es inactivar la función de la enzima anómala uniéndose a su sitio activo y provocando la muerte únicamente de las células que tienen el cromosoma Filadelfia.
Este fármaco no se diseñó por computadora, sino a fuerza de ensayo y error. En 1976 se aisló la estaurosporina, una molécula producida por bacterias marinas que inhibe la actividad de las cinasas, matando cualquier tipo de célula. Partiendo de allí, el equipo del bioquímico Nicholas B. Lydon, en Suiza, identificó una estructura similar, pero más simple y específica para inhibir una clase de cinasas. Con esa molécula base, los investigadores diseñaron cientos de moléculas con modificaciones estructurales que se fueron probando una a una. Fue hasta 1996 cuando, en colaboración con el oncólogo Brian Druker, del Instituto Dana-Farber, en Boston, se encontró entre todas esas moléculas una que inhibía exclusivamente la acción de la fusión de los cromosomas 9 y 22. Finalm ente, en 2001 el imatinib se aprobó como tratamiento para la leucemia mieloide crónica. Así, una enfermedad mortal se transformó en un padecimiento crónico y tratable. Pero probar cada molécula fue un proceso muy largo y costoso. Los estudios in silico agilizan este trabajo: es algo parecido a encontrar entre cientos de llaves (los ligandos) la que abre una puerta, pero en lugar de probar una por una, la simulación en computadora nos indica cuáles tienen las mayores probabilidades de funcionar.
Unirse o no unirse, esa es la cuestión
Una de las estrategias usadas para evaluar si un ligando posee alguna propiedad farmacológica es determinar si puede unirse a una proteína objetivo en la célula. La herramienta más utilizada para esto es el docking, o acoplamiento molecular, que no solo nos dice si el ligando y la proteína son compatibles, también predice la fuerza de unión y la variante del ligando que encaja mejor con la proteína objetivo. Normalmente, la fuerza de unión determinará la duración del efecto del nuevo fármaco: entre más fuerte la unión, más duradero el efecto.
La forma en que un ligando activa o inhibe la función de su receptor se denomina conformación bioactiva. En el docking rígido, la proteína se mantiene en una sola posición geométrica mientras el ligando se mueve hasta encontrar un modo de unión de alta afinidad. En el docking flexible, la proteína se somete a efectos dinámicos hasta encontrar la conformación bioactiva del ligando. Es un poco como esos juguetes para bebés que consisten en un cubo con perforaciones de diversas formas en las que hay que encajar la pieza de la forma correspondiente. El docking rígido equivaldría a buscar piezas que quepan en un hueco específico y el flexible a moldear la pieza hasta encontrar la forma que mejor encaje en el hueco. La computadora asigna una puntuación a cada conformación (o forma geométrica) del ligando. Las más afines a la proteína objetivo, las más estables y que requieran menos energía para unirse, serán las mejor calificadas.
No es lo mismo, pero es igual
A pesar de que el imatinib cambió el pronóstico de la leucemia mieloide crónica, algunos pacientes desarrollaron resistencia al tratamiento. Mutaciones en el gen que hace que la célula fabrique la proteína BCR-ABL modificaron el sitio activo de la proteína original, debilitando la unión del imatinib. A partir de la estructura cristalizada del imatinib y el receptor unidos, se hicieron modificaciones al esqueleto del fármaco, añadiendo o cambiando átomos que reforzaban la unión. Los avatares de estas nuevas moléculas fueron evaluados por docking molecular y así se encontró un nuevo fármaco más potente y eficaz para tratar esa leucemia: el nilotinib. En este ejemplo, los investigadores tomaron una molécula base y suponiendo que la estructura es la clave de la eficacia, crearon en el laboratorio versiones ligeramente modificadas que tenían un efecto igual o mayor.
Pero toda regla tiene excepciones: hay compuestos que, sin parecerse en estructura, tienen efectos similares, por ejemplo, el ketorolaco y el ibuprofeno, dos medicamentos antiinfl amatorios y analgésicos. También hay moléculas que se parecen, pero cuyos efectos son opuestos, como la psilocibina y la clozapina: la primera es un alucinógeno, la segunda un antipsicótico.
Volvamos al ejemplo del juguete. En la abertura cuadrada cabría perfectamente un triángulo si su altura es menor que el lado del cuadrado.
Pero el triángulo no toca todos los bordes del espacio cuadrado; en otras palabras, interactúa de forma diferente. Lo mismo ocurre entre los fármacos y su receptor: los átomos de ambos interactúan entre sí para desencadenar un efecto. Si una molécula no interactúa con los mismos aminoácidos (moléculas que se combinan para formar proteínas) o interactúa con otros, probablemente el efecto será menor o hasta opuesto.
El docking solamente indica si la unión entre dos moléculas será favorable o no, pero hay otras características importantes que determinan la actividad biológica del ligando.
¿Y la inteligencia artificial?
ChatGPT es un modelo de lenguaje de inteligencia artificial (IA) que simula conversaciones con los usuarios y responde todo tipo de preguntas. Le preguntamos si la IA puede ayudar al diseño de fármacos y si nos podía dar algún ejemplo. Ésta fue su respuesta:
“La IA puede predecir la actividad biológica de las moléculas candidatas a fármacos mediante el uso de modelos de aprendizaje automático (machine learning). Estos modelos se entrenan con grandes conjuntos de datos de experimentos biológicos y clínicos que incluyen información sobre la estructura de las moléculas y su actividad biológica. Una vez entrenado, el modelo puede utilizarse para predecir la actividad biológica de nuevas moléculas.
Por ejemplo, se podría entrenar un modelo de aprendizaje profundo en datos sobre la actividad biológica de una amplia variedad de moléculas antivirales. Una vez entrenado, el modelo podría utilizarse para predecir la actividad antiviral de nuevas moléculas candidatas. De esta manera, los investigadores podrían centrarse en las moléculas más prometedoras para su posterior evaluación en experimentos biológicos y clínicos.”
Prohibido dejarlo a la suerte
Para que un ligando sea clínicamente útil, en su diseño hay que tomar en cuenta otras características, pues aunque la nueva molécula tenga buenas probabilidades de unirse al objetivo puede resultar altamente tóxica, degradarse muy rápido en el organismo e incluso ser imposible de absorber.
Para superar estos obstáculos, otros métodos in silico calculan las propiedades fisicoquímicas, tóxicas y hasta la dosis en que las moléculas recién diseñadas ejercerán su acción. Un ejemplo es el método denominado “relación cuantitativa estructura- actividad” (QSAR, por sus siglas en inglés), que da una aproximación de estas propiedades comparando el ligando con fármacos conocidos de estructura y características similares a él. Con ello construye el modelo matemático en el que se basarán sus predicciones. Como estos modelos dan una idea de las propiedades del nuevo compuesto, se reduce la cantidad de experimentos y animales necesarios para ponerlo a prueba.
Los métodos in silico permiten filtrar una lista de candidatos a moléculas con acción farmacológica para solamente sintetizar las más prometedoras.
La magia de crear
En el laboratorio materializamos los avatares de las moléculas mejor calificadas por los métodos in silico. Para sintetizarlos se hacen reaccionar materias primas hasta que forman la estructura deseada.
Si bien a la hora de crear in silico el límite es la imaginación, muchas veces las rutas de síntesis química no son sencillas. Algunas requieren pasar de una reacción a otra para obtener productos intermedios que nos permitan continuar el camino. Por eso es necesario buscar la ruta óptima que maximice la producción con el fin de reducir el costo del producto final.
Los obstáculos no acaban ahí. A veces las nuevas moléculas resultan químicamente inestables en condiciones ambientales y una vez purificadas se degradan, lo que causa no pocas frustraciones. Pero es fascinante que cambiar un solo átomo por otro, o simplemente modificar su posición, pueda dar un efecto completamente diferente.
De la computadora a la farmacia
Una vez sintetizada una nueva molécula, hay que probar su efecto en un sistema biológico: en ensayos preclínicos en células (in vitro) y animales (in vivo), y ensayos clínicos en humanos. Desarrollar un nuevo fármaco desde su diseño hasta su aprobación toma entre 10 y 15 años, un proceso de gran costo económico y humano, si además se toma en cuenta que solo se aprueba el 12 % de los fármacos que llegan a la fase clínica.
Aunque los resultados de los modelos computacionales solo son simulaciones —avatares de estructuras químicas que predicen cómo actuarían en la realidad—, el diseño de fármacos in silico gana cada día más terreno por ser una manera más rápida y certera de encontrar nuevos medicamentos con efectos específicos, menos tóxicos para el paciente, a menor costo e incluso menos dañinos para el ambiente.
Agradecemos al Dr. Jonathan Cueto Escobedo, al M. en C. Luis Ángel Luis Gil y a la Dra. Miríam Guadalupe Báez por sus aportaciones y comentarios, que enriquecieron este artículo.
- Prieto Martínez, Fernando y José L. Medina Franco, “Diseño de fármacos asistido por computadora: cuando la informática, la química y el arte se encuentran”, TIP. Revista especializada en ciencias químico-biológicas, México, 2020: >https://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S1405-888X2018000200201.
- Scior, Thomas, Evelyn Martínez Morales y Eduardo Salinas Stefanón, “Los modelos in silico, una herramienta para el conocimiento farmacológico”, Elementos: Ciencia y Cultura, Benemérita Universidad Autónoma de Puebla, México, 2007: https://elementos.buap.mx/index.php.
Los autores son investigadores del Laboratorio de Diseño y Desarrollo de Nuevos Fármacos e Innovación Biotecnológica de la Escuela Superior de Medicina del Instituto Politécnico Nacional (IPN).
José Correa Basurto es médico, maestro en farmacología y doctor en investigación en medicina. Ha sido responsable técnico de proyectos nacionales e internacionales de diseño y desarrollo de fármacos y nanovacunas.
Humberto L. Mendoza Figueroa es doctor en farmacología. Desarrolla proyectos relacionados con la química medicinal para el diseño y síntesis química de nuevas moléculas con actividad anticancerígena.
Cynthia Fernández Pomares es investigadora del programa Estancias Posdoctorales por México 2021 del Consejo Nacional de Ciencia y Tecnología (Conacyt). Estudia el efecto de compuestos anticancerígenos sobre el metabolismo.















